王恩多研究组发现茎接触折叠调控亮氨酰-tRNA合成酶的氨基酰化与编校活力
来源:
时间:2015-05-25
3月27日,国际学术期刊The Journal of Biological Chemistry在线发表了中国科学院上海生命科学研究院生物化学与细胞生物学研究所王恩多研究组题为"Modulation of aminoacylation and editing properties of leucyl-tRNA synthetase by a conserved structural module"的最新研究成果。
亮氨酰-tRNA合成酶(LeuRS)在体内负责催化亮氨酸和对应亮氨酸tRNA (tRNALeu)之间的酯化反应(氨基酰化反应),生成亮氨酰-tRNA(Leu-tRNALeu), 为蛋白质生物合成提供原料。根据进化融合假说,最初的LeuRS仅有催化活性中心的核心结构域,在进化过程中逐渐添加负责编校的CP1编校结构域,参与结合tRNA的反密码子结合结构域及C-末端结合结构域,稍欠保守的亮氨酸专一结构域(LSD)等。有人报道茎接触折叠(stem contact fold, SCF)参与连接LeuRS催化活性中心KMSKS环与反密码子结合结构域并呈现保守β-α-α-β-α的拓扑结构,该结构域广泛存在于Ia及Ib类氨基酰-tRNA合成酶中。然而关于此结构域的功能尚未见报道。
王恩多研究组的闫卫博士等系统地研究了来自原核生物、古菌及真核生物LeuRS位于SCF结构域的重要氨基酸残基,发现第3个α螺旋(SCF-α3)保守的碱性氨基酸及甘氨酸残基调控LeuRS的氨基酰化与编校活力,这些氨基酸残基的突变降低了酶与tRNA
Leu的亲和力。进一步的研究发现这些碱性氨基酸残基参与识别
tRNALeu反密码子茎的核苷酸。
另外,SCF中第二个β-折叠 (SCF-β)的酸性氨基酸残基参与LeuRS依赖tRNA的编校反应并识别tRNA
Leu的D环大小。这些发现充分证明了SCF 参与LeuRS氨基酰化及编校活力的调节。序列比对发现天然缺失编校结构域的运动型支原体LeuRS(
MmLeuRS)的SCF缺少重要的甘氨酸残基及酸性氨基酸残基。此前,王恩多研究组曾通过向天然无编校结构域的
MmLeuRS嵌合
EcLeuRS的CP1,LSD结构域,构建了嵌合酶
MmLeuRS-CP1/LSD(MEL)。
本研究中将鉴定到的SCF重要元件嵌合至MEL的SCF-β,得到的嵌合酶增强了对误氨基酰-MmtRNALeu(CAA)等受体的编校活力。另一方面,MmtRNALeu(CAA)自身D环增大也增强了嵌合酶MEL的编校活力。随着SCF重要氨基酸残基的替换, MmLeuRS逐步恢复了对误氨基酰化产物水解的功能,同时构建的嵌合酶MEL成为氨基酰化与编校功能较强的功能型酶,在一定程度上人工模拟了LeuRS的进化。研究也证明了LeuRS在长期进化中,其SCF通过调整自身序列来配合CP1结构域的嵌合,辅助它更好地水解错误产物,大大加深了人们对SCF结构域功能的认识,暗示除LeuRS的合成结构域和编校结构域外,SCF也是设计针对病原菌新药的靶结构。
该项研究获得国家基础研究基金、国家自然科学基金、中科院、上海市科委等的经费资助。
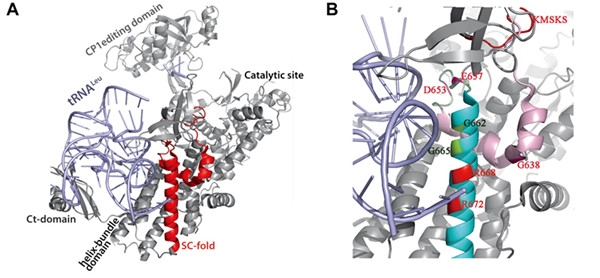
图示:E. coli LeuRS晶体结构示意图及其茎接触折叠重要氨基酸残基
