5月3日,国际学术期刊《科学报道》Scientific Reports在线发表了中国科学院上海生命科学研究院生物化学与细胞生物学研究所国家蛋白质科学中心(上海)黄超兰研究团队与复旦大学杨芃原研究团队、中科院计算所贺思敏研究组的合作研究论文“pGlyco: a pipeline for the identification of intact N-glycopeptides by using HCD-and CID-MS/MS and MS3”。该研究报道了一款解析完整 N-糖肽质谱数据的软件——pGlyco。该软件利用了质谱采集数据的HCD (High Collsion dissociation)和CID(Collision-induced dissociation )碎片信息实现了糖肽的鉴定解析。pGlyco软件填补了目前完整N-糖肽鉴定方法中缺少false discovery rate (FDR)分析、糖肽鉴定方法中缺少充足碎片信息的空缺,挑战了糖蛋白组学分析中最困难的方面,解决了目前N-糖肽鉴定中FDR高等问题,达到对蛋白糖基化微异质性的可靠表征。
糖蛋白对人体维持正常的生理功能发挥举足轻重的作用,但糖蛋白的鉴定目前仍是蛋白质组学中最难的部分。组成糖蛋白的糖原类型多样、糖链结构复杂等原因是阻碍糖蛋白组学研究的主要障碍。传统的糖肽鉴定的方法分为糖原鉴定和肽段骨架鉴定两步。近年来,科学家们研发了多种质谱技术和解读完整糖肽的生物信息工具。一种方法是利用CID-MS/MS结合ETD-MS/MS或靶向 MS3技术直接解读完整糖肽的质谱数据。另一种比较流行的质谱方法是利用HCD-子离子-ETD(HCD-pd-ETD)方法鉴定完整糖肽。然而,这些方法鉴定到的糖肽即使在肽段-谱图匹配分数高时仍会发生FDR高的情况。由上述三家单位合作研发的pGlyco软件能解决糖组学研究中最难的问题:糖肽骨架鉴定和高FDR。pGlyco具有以下特点:1)利用来自于HCD-MS/MS和CID-MS/MS两种互补的碎片信息鉴定糖原;2)引入一个全新target-decoy方法计算糖原的FDR;3)利用HCD-MS/MS产生的响应最高的质谱峰进一步进行数据依赖采集MS3信息,对糖肽进行鉴定。通过整合上述三个特点,pGlyco将能用详细的质谱图谱信息同时鉴定糖肽和肽段。
该研究的质谱分析工作获得了国家蛋白质科学研究(上海)设施质谱分析系统彭超博士的协助和技术支持。质谱分析系统自2014年5月开放至今,累计服务用户课题超过250个,为多项国家科技战略先导专项、973和863项目等提供科研保障和技术支撑。截至2016年4月,质谱分析系统用户已在Science、Nature、Nature子刊、Mol Cell、PNAS、JACS等国际著名期刊上发表文章10多篇,并有2篇共同通讯作者文章分别在EMBOJ和Scientific Report上,为各领域科学研究起到了重要的推动作用。
该研究工作得到中国国家基础科学研究基金(973专项)、国家自然科学基金、中国高科技研究和发展项目(863项目)等的经费资助。
文章链接:http://www.nature.com/articles/srep25102
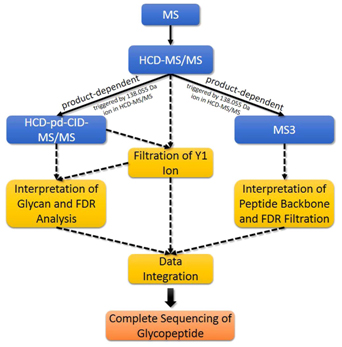
图1. pGlyco的工作流程。首先样品用HCD-MS/MS的方法分析(NCE=40%), 然后分别进行子离子依赖的CID-MS/MS与数据依赖的MS3的数据采集。pGlyco 通过整合三种互补的质谱数据所包含的独特信息,这些信息基于肽段骨架质量数和保留时间。实线表示数据采集,虚线表示数据解读。