10月24日,国际知名学术期刊Nature Structural & Molecular Biology在线发表了中国科学院上海生命科学研究院生物化学与细胞生物学研究所国家蛋白质科学中心(上海)丛尧研究组的研究论文“Staggered ATP binding mechanism of eukaryotic chaperonin TRiC (CCT) revealed through high-resolution cryo-EM”。该研究应用冷冻电镜技术结合生化分析揭示了真核生物分子伴侣素TRiC(又名CCT)在结合ATP过程中按特定的时空顺序、分阶段进行的结构机制,实现了对TRiC开环状态的亚基精确定位,为深入阐释TRiC高度协同性及其特异性识别并折叠底物的机制提供了结构基础。
细胞中蛋白质的正确折叠对其发挥正常的生物学功能、维持细胞内环境的稳态具有重要作用。真核细胞中的分子伴侣素TRiC可以协助~10%的胞质蛋白正确折叠,其底物包括许多重要的结构性和调节性蛋白,如细胞骨架蛋白的主要成分肌动蛋白(actin)和微管蛋白(tubulin),肿瘤抑制蛋白VHL和p53,及引起亨廷顿氏舞蹈病的蛋白Huntingtin(Htt)等。TRiC功能的缺失与癌症和神经退行性疾病等人类疾病密切相关。TRiC 拥有8个不同的亚基,分子量达到1 MDa,是最为复杂的分子伴侣素,其功能的实施与其在ATP 驱动下的构象变化密切相关。然而由于该超大分子复合体具有动态性、多构象性、溶液中取向分布倾向性等特征,至今仍缺乏其高分辨率结构信息,其亚基排布仍存在争议,限制了人们对TRiC协助底物折叠机制的认知。
丛尧研究员带领博士研究生臧云祥、金明梁及硕士研究生王胡萍等实验室成员,依托国家蛋白质科学中心(上海)的先进冷冻电镜设施,解析了酵母源的TRiC在首次发现的 nucleotide partially preloaded(NPP)状态下及AMP-PNP模拟的ATP结合状态下的高分辨冷冻电镜三维结构(分别达到4.7 Å及4.6 Å分辨率),其中ATP结合状态结构是目前TRiC在开环状态下分辨率最高的三维结构。同时,通过发展的亚基中位eGFP标签定位方法,实现了对开环状态下TRiC亚基的精确定位。观测到CCT2亚基在NPP状态下存在特殊构象,揭示了CCT2亚基在整个复合物协同变构过程中的关键作用。首次发现TRiC位于CCT6一侧的三个亚基可以锁住酵母本底环境中的核苷酸(主要以ADP形式存在)并且不易被取代,揭示了TRiC在结合ATP过程中按特定的时空顺序、分阶段进行的结构机制。研究阐释了TRiC复合体在进化过程中在结构和功能上已分化为两个部分,以及TRiC如何协调其构象变化产生的机械力进而实现协助底物折叠为最终产物的结构基础,加深了人们对TRiC高度协同性及其特异性识别并折叠底物机制的认知。
该研究得到国家蛋白质科学中心(上海)冷冻电镜系统,数据库与计算分析系统、质谱系统、核磁系统、显微镜系统与规模化蛋白质制备系统的大力支持。该研究得到生化细胞所周金秋研究员和周兆才研究员,美国贝勒医学院Wah Chiu教授的大力支持,并得到国家自然科学基金委、国家科技部、中国科学院战略性先导科技专项(B类)和上海市科委等的资助。
原文链接:http://www.nature.com/nsmb/journal/vaop/ncurrent/full/nsmb.3309.html
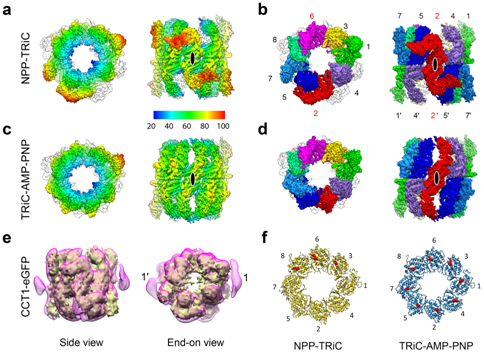
图1、 TRiC在NPP和ATP结合状态的冷冻电镜结构(a)NPP-TRiC的顶视图与侧视图,颜色随半径增大渐变。(b)NPP-TRiC的顶视图与侧视图及亚基排布。八个不同亚基由不同的颜色表示。(c-d)TRiC-AMP-PNP冷冻电镜结构。(e)通过自行发展的亚基中位eGFP标签定位方法,实现对开环状态下CCT1亚基的精确定位。(f)NPP-TRiC与TRiC-AMP-PNP状态下的核苷酸分布图。