2月2日,中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)丁建平研究组在Nature Communications发表题为“Molecular mechanism for vitamin C-derived C5-glyceryl-methylcytosine DNA modification catalyzed by algal TET homologue CMD1”的论文,该研究揭示了衣藻中TET同源蛋白CMD1以维生素C作为共底物(co-substrate)催化DNA中5mC修饰形成5gmC修饰的分子机制。
近年来,表观遗传学研究领域一直致力于发现新型DNA修饰方式。研究表明,TET蛋白利用Fe2+和2-酮戊二酸(2-oxogluatarate, 2-OG)能够催化5mC发生多步氧化反应,依次形成5hmC、5fC以及5caC。2019年,徐国良院士等多个课题组合作研究发现莱茵衣藻(Chlamydomonas reinhardtii)中TET同源蛋白CMD1(5-methylcytosine modifying enzyme 1)能够以维生素C(vitamin C, VC)为共反应底物,催化DNA的5mC产生一种全新的DNA修饰5gmC(5-glyceryl-methylcytosine),并在莱茵衣藻的光合作用中发挥重要调节作用。CMD1作为2-OG依赖的双加氧酶家族成员,催化5mC形成5gmC需要VC而非2-OG,这与该家族其他成员显著不同。在CMD1被报道之前,VC主要作为一种抗氧化剂发挥功能,或作为还原剂促进2-OG依赖的双加氧酶的催化活性,未有研究表明VC能够作为共反应底物发挥生物学功能。CMD1如何利用VC而非2-OG催化5mC发生修饰,以及CMD1是否具有底物特异性等科学问题及其分子机制都不清楚。
丁建平研究组的助理研究员李文婧、研究员张天龙和博士研究生孙明亮合作对CMD1的结构、功能和分子机制开展了深入研究。他们基于生化分析发现,CMD1对不同长度、结构及5mC修饰水平的DNA底物具有相近的亲和力,但对含有5mCpG的DNA有一定程度的底物偏好性。他们解析了CMD1原酶、结合VC、结合DNA或5mC-DNA、以及结合5mC-DNA和VC的复合物的高分辨率晶体结构。 结构分析表明,CMD1的整体结构采用典型的2-OG依赖的双加氧酶家族的DSBH(double-stranded β-helix)折叠方式。在CMD1结合VC以及结合5mC-DNA和VC的复合物结构中,VC均以内酯形式存在。相互作用分析表明,VC通过广泛的氢键和疏水相互作用结合在CMD1的活性位点,并通过单配位与Fe2+螯合,这与其他2-OG依赖的双加氧酶中2-OG以双配位与Fe2+螯合的方式不同。体外酶活实验表明,CMD1催化的反应只有当VC以内酯形式存在时才能发生。CMD1-VC-5mC-DNA三元复合物的结构代表了CMD1催化反应的起始状态,在该结构中DNA底物主要通过磷酸骨架与CMD1正电荷富集的表面相互作用,DNA底物链中的5mC从双链DNA中翻转出来并插入CMD1的活性位点;5mC的甲基基团指向VC,但没有被特异性识别,因此未修饰的C也能够插入CMD1的活性位点。突变体实验表明,CMD1的催化活性依赖于金属离子、VC和DNA底物的正确结合。与TET蛋白的结构比较阐明了CMD1和TET蛋白使用不同共反应底物催化反应的结构基础,但CMD1和TET蛋白对底物5mC的结合和识别方式却非常相似。这些研究结果揭示了CMD1以VC为共底物催化DNA中5mC修饰形成5gmC修饰的分子机制。
分子细胞卓越中心徐国良研究组参与了这项工作。该研究工作得到了上海同步辐射光源BL17U1线站和国家蛋白质科学研究设施(上海)BL18U和BL19U1线站、分子细胞卓越中心分子平台的大力支持,以及中科院先导B项目和科技部研发专项的经费支持。
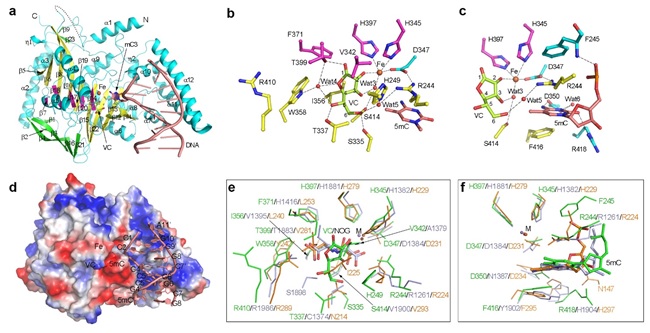
(a) CMD1-VC-5mC-DNA三元复合物的整体结构。(b, c) CMD1的活性位点。(d) CMD1的表面电势分布以及与5mc-DNA的结合方式。(e, f) CMD1与TET蛋白活性位点的结构比较(绿色:CMD1;浅蓝色:HsTET2;橙色:NgTET1)。