2月1日,国际学术期刊Nucleic Acids Research在线发表了中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)周小龙组与王恩多组最新合作研究成果“Commonality and diversity in tRNA substrate recognition in t6A biogenesis by eukaryotic KEOPSs”。
在细胞内所有RNA分子中,tRNA含有最为密集且种类最为多样的转录后修饰。截止目前,已经发现约120种左右的tRNA转录后修饰。tRNA修饰可以稳定tRNA的二级与高级结构,保证密码子-反密码子配对的精确性,促进mRNA翻译的保真性和效率等。其中,若干tRNA修饰(包括第37位腺嘌呤N6-苏氨酰基甲腺苷酸, t6A37)是三界生物共有的、且为细胞生命活动所必须的修饰。在少数极端生物中,多数tRNA非必须,但两种修饰必不可少,即t6A37以及m1G37,突显了t6A37在mRNA翻译及细胞正常生命活动中的关键作用。t6A修饰位于负责解码ANN (N = A, T, G, C)密码子的tRNA 第37位腺嘌呤。负责t6A修饰的基因在三界生物中已被鉴定,其生物学功能在细菌和古菌中研究较多,但对于人细胞t6A修饰的分子机制及功能的认识非常有限。真核生物细胞质中,YRDC以及5元复合物KEOPS (由OSGEP, TP53RK, TPRKB, LAGE3及GON7组成)负责细胞质tRNA的t6A修饰。这6种基因中的每一个基因发生的突变会使t6A修饰缺陷,均导致Galloway-Mowat 综合症,但致病机制不详。
该研究中,研究人员从人细胞中纯化了14种解码ANN密码子的细胞质tRNA,UPLC-MS/MS鉴定这14种tRNA均含有t6A修饰;进一步用来源于酿酒酵母、人与线虫细胞质的KEOPS复合物开展t6A修饰活力测定,揭示了t6A修饰机器与起始tRNAMet (tRNAMeti)存在着协同进化,即细菌及单细胞真核生物(酿酒酵母等)而非多细胞真核生物(人及线虫等)t6A修饰机器利用C33作为反向识别元件,进而在多细胞真核生物中进化出含有C33的tRNAMeti;系统揭示了人KEOPS精确性识别tRNA底物需要特定的tRNA元件,包括C32以及D茎中的两对保守的碱基对,但通用的CCA末端在t6A修饰中的功能因物种而异,具有多样性;阐释了人KEOPS主要定位于细胞质,只能在没有内含子的成熟tRNA上介导t6A修饰;进一步构建了催化亚基OSGEP的敲低细胞系,发现t6A修饰缺陷不影响所有tRNA的稳态水平,也不影响绝大多数tRNA的氨基酰化水平,但体内与体外实验均证明,t6A修饰是细胞质异亮氨酰-tRNA合成酶(hIleRS)识别tRNAIle的关键识别元件;通过构建mRNA翻译的荧光报告系统,证明t6A修饰对于密码子-反密码子的精确配对具有重要作用,阻止了+1移码突变。
该研究明确人所有的ANN解码tRNA均具有t6A修饰,发现t6A修饰机器与tRNAMeti的协同进化,首次阐明真核生物KEOPS识别tRNA底物的分子机制,揭示t6A修饰是hIleRS识别tRNAIle的关键识别元件并进一步阐释了t6A修饰在mRNA翻译中的关键作用。这些研究结果为认识人细胞质tRNA t6A修饰的分子机制、揭示修饰酶及tRNA突变相关人类疾病的发生机制提供了理论基础。
分子细胞卓越中心博士研究生王金涛与周敬波为本文共同第一作者,周小龙研究员与王恩多研究员为本文共同通讯作者。兰州大学张文华教授、中国药科大学陈美容教授参与了本研究。感谢分子细胞卓越中心分子生物学技术平台在质谱分析方面提供的帮助。该研究得到国家重点研发计划、基金委以及中科院的经费资助。
文章链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkac056/6519365?login=true
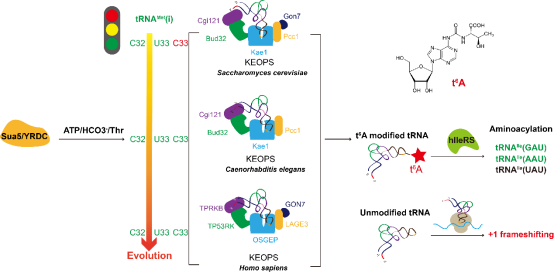
真核KEOPS复合物和起始tRNAMet的协同进化及其细胞功能