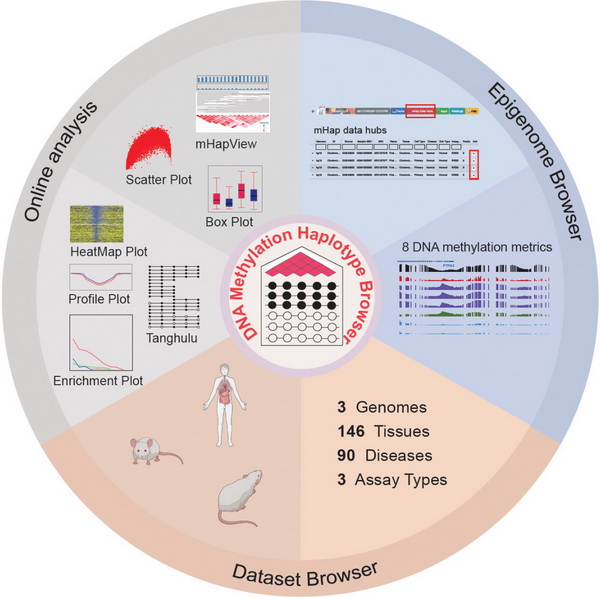
10月13日,国际学术期刊Nucleic Acid Research在线发表了中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)石建涛研究组与上海交通大学合作的最新研究成果“mHapBrowser: a comprehensive database for visualization and analysis of DNA methylation haplotypes”。这项研究工作构建了一个全新的、开源的、易于科研数据共享的DNA甲基化单体数据库— mHapBrowser (http://mhap.sibcb.ac.cn),并且能够支持在线运算和可视化。
DNA甲基化是一种重要的表观遗传修饰,在基因调控、基因组稳定性、细胞分化和疾病发生中起关键作用。在人类基因组中,DNA甲基化的动态变化主要发生在CpG位点上。基于亚硫酸盐测序的方法,如WGBS、RRBS和靶向BS-seq,能够在单核苷酸分辨率检测DNA甲基化。深入挖掘DNA甲基化的功能需要数据库和相关分析工具的支持。目前已经存在一些DNA甲基化相关数据库,如MethDB、MethBase、DNMIVD、NGSmethDB和Meth-Bank,但这些数据库主要关注每个CpG位点的平均甲基化水平,而忽略了同一条染色体上的甲基化模式(DNA甲基化单体)。
在前期工作中,石建涛研究组定义了一种新的数据格式mHap来存储DNA甲基化单体信息,该格式相比BAM文件实现了40-150倍的存储压缩,同时完整保留了CpG位点的甲基化信息。基于mHap格式,该团队还合作开发了DNA甲基化单体分析工具mHapTk,可以方便计算多个mHap的统计量如PDR、CHALM、MCR、MHL等以描述DNA甲基化特征。甲基化单体存储格式和分析工具的开发为后续数据库的构建奠定了基础。
mHapBrowser数据库收录了人类、小鼠和大鼠三个物种的数据,分别为5808个人类样本、490个小鼠样本和69个大鼠样本。每个样本都包含丰富的元数据信息,如GEO/SRX编号、组织来源、疾病状态、比对率、覆盖度和平均甲基化等,方便用户筛选数据。数据库集成了WashU Epigenome浏览器,可以直接展示每个样本在全基因组尺度上的mHap统计量,包括平均甲基化、PDR、CHALM、MCR、MBS、MHL、Entropy和连锁不平衡系数R2。数据库还提供在线分析服务,可以方便地比较不同样本之间的mHap特征。
分子细胞卓越中心研究生洪毓阳、研究助理刘磊琴、研究生丰琰以及上海交通大学博士生张志强为本研究的共同第一作者。分子细胞卓越中心石建涛研究员和上海交通大学医学院附属仁济医院许琼主治医师为本文的共同通讯作者。该数据库的搭建工作得到分子细胞卓越中心高性能计算平台的大力协助。该项目由国家自然科学基金资助。
文章链接:https://doi.org/10.1093/nar/gkad881