9月8日,国际学术期刊Advanced Science发表了中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)吴薇研究组的研究成果:“Benchmarking Ploidy Estimation Methods for Bulk and Single-Cell Whole Genome Sequencing”。 该研究首次系统评估了19种倍性检测算法在肿瘤基因组分析中的性能,为癌症临床诊断和基础研究提供了工具选择的关键标准。
染色体数量变异引起的细胞倍性异常是癌症的重要标志,与肿瘤演进和患者预后密切相关。尽管已有多种基于全基因组测序的倍性检测工具,但其性能缺乏统一评估。该研究通过整合实验数据集和涵盖不同测序平台、肿瘤纯度梯度及测序深度的模拟数据集,对11种常规WGS工具和8种单细胞工具进行了全面测试。研究发现,在肿瘤纯度大于30%的常规样本中,算法PURPLE展现出最优性能,即使在10X低深度测序条件下仍保持稳定;而对于低纯度样本,PyLOH在纯度评估中表现突出但倍性检测能力有限。在单细胞测序领域,SeCNV算法在肿瘤细胞倍性识别中准确率最高。值得注意的是,所有现有工具均无法准确识别全基因组加倍所引起的异常整倍体细胞,且难以适配长读长测序数据。
该研究进一步揭示了工具性能的关键影响因素:数据预处理流程可提升TitanCNA等工具的纯度评估精度;基于Strelka的突变检测策略在保证准确性的同时将效率提升17倍;测序深度对Accucopy等工具有显著制约。这些发现为肿瘤基因组异质性研究、癌症预后标记物开发及个体化治疗策略提供了方法学支撑。
分子细胞卓越中心博士研究生宋亚伟、梅自律为论文共同第一作者,吴薇研究员与吴树恒博士为共同通讯作者。研究工作获得国家重点研发计划、中国科学院战略先导专项、中国科学院国际伙伴计划、国家自然科学基金、上海市启明星项目及上海市科技重大专项的支持,并得到分子细胞卓越中心高性能计算平台的技术支撑。
文章链接:https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202507839
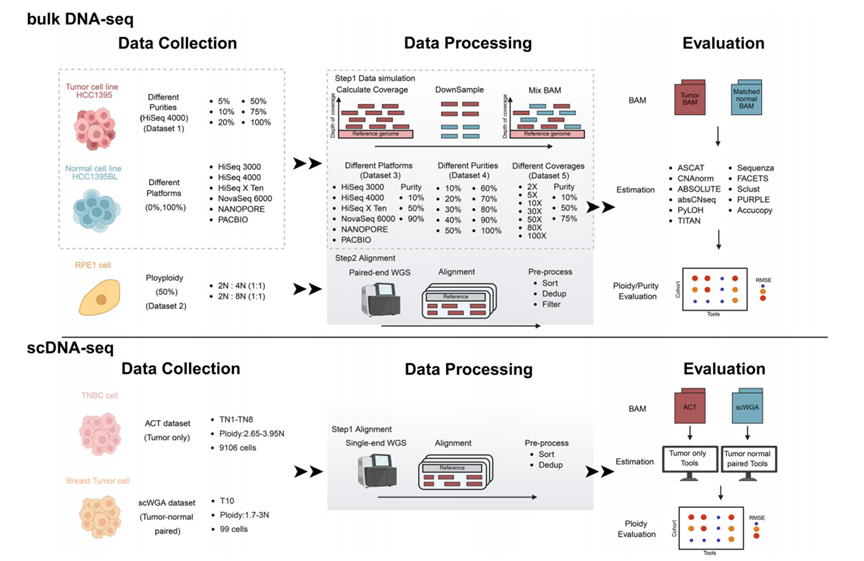
系统评估工作流程示意图