12月8日,国际学术期刊Genome Research在线发表了中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)石建涛研究员与上海交通大学医学院附属瑞金医院方海教授的最新研究成果“A DNA methylation haplotype block landscape in human tissues and preimplantation embryos reveals regulatory elements defined by comethylation patterns”。该研究收集了17种正常组织的全基因组DNA甲基化数据(WGBS),并鉴定出58,385个DNA甲基化单体水平上的共甲基化区间(MHB)。大规模数据整合分析表明,MHB富集在组织特异性增强子区域,并和基因的激活相关,是一种由DNA甲基化单体特征定义的转录调控元件。
DNA甲基化以及相关的调控元件在基因表达调控中发挥着重要的作用。传统的方法大多利用平均甲基化来定义DNA甲基化相关的特征区域,比如UMR、LMR、PMD等 (Burger et al., 2013)。然而,这些方法只关注CpG位点的平均甲基化水平,而忽略了同一条染色体上的甲基化修饰模式(DNA甲基化单体)。相同的平均甲基化水平可能对应完全不同的DNA甲基化单体特征,因此具有不同的调控功能。此外,DNA甲基化单体上的两个CpG位点之间的甲基化状态往往不是独立的,可能存在显著的关联性。在前期的工作中,研究者利用遗传学上连锁不平衡的方法来识别共甲基化区域,并把它们命名为DNA甲基化单体区块(MHB)(Shoemaker et al., 2010;Guo et al., 2017)。MHB作为生物标志物,在肿瘤早筛领域有较大的应用前景(Wu et al., 2022),然而它们的组织特异性以及潜在的生物学功能仍然不明确。
在以往的研究中,石建涛研究组定义了一种新的DNA甲基化单体格式mHap(Zhang et al., 2021),并开发了相应的分析工具mHapTk(Ding et al., 2022)和数据库mHapBrowser(Hong et al., 2023),使得大规模、高效的整合分析成为可能。本研究收集了72个WGBS样本数据,构建了17种正常组织的DNA甲基化单体图谱,鉴定了每种组织中的MHB及相关的基因组特征。在矫正了平均甲基化的情况下,MHB比UMR、LMR更加富集染色质开放区域,揭示MHB具有潜在的转录调控功能。整合ENCODE和Roadmap数据发现,MHB富集组织特异性的增强子,包括超级增强子。进一步分析发现,MHB倾向于分布在组织特性基因周围,和基因的激活密切相关。在两种组织中,即使平均甲基化相同的基因,MHB的获得或者丢失也和差异基因表达显著性相关。以上结果都显示,MHB是一种由DNA甲基化单体特征定义的基因转录调控元件。
分子细胞卓越中心研究生丰琰以及上海交通大学博士生张志强为该论文的共同第一作者。分子细胞卓越中心石建涛研究员和上海交通大学医学院附属瑞金医院方海教授为该论文的共同通讯作者。该项研究得到了分子细胞卓越中心高性能计算平台的大力支持。该项目由国家自然科学基金资助。
文章链接:https://genome.cshlp.org/content/33/12/2041.abstract
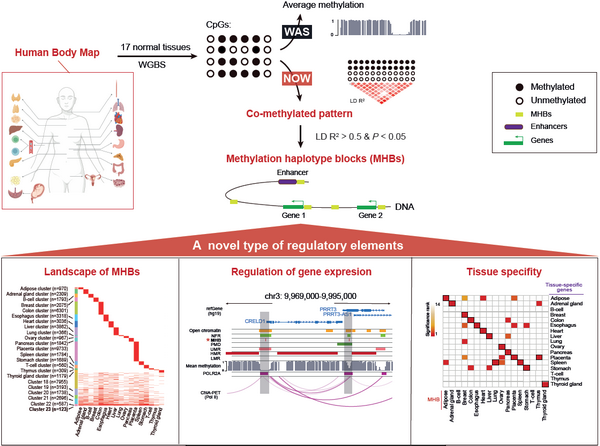
正常组织共甲基化区间的鉴定